black-forest-labs/flux



flux 是一个用于运行 FLUX.1 模型进行图像生成和编辑的官方推理库。
- 提供多种模型,包括文本到图像、填充、结构条件等功能。
- 支持本地安装和使用,方便用户快速上手。
- 提供易于使用的 Python 接口以及命令行工具,简化 API 调用过程。
- 详细文档可帮助用户了解如何有效利用 API。
QwenLM/Qwen2.5-Coder


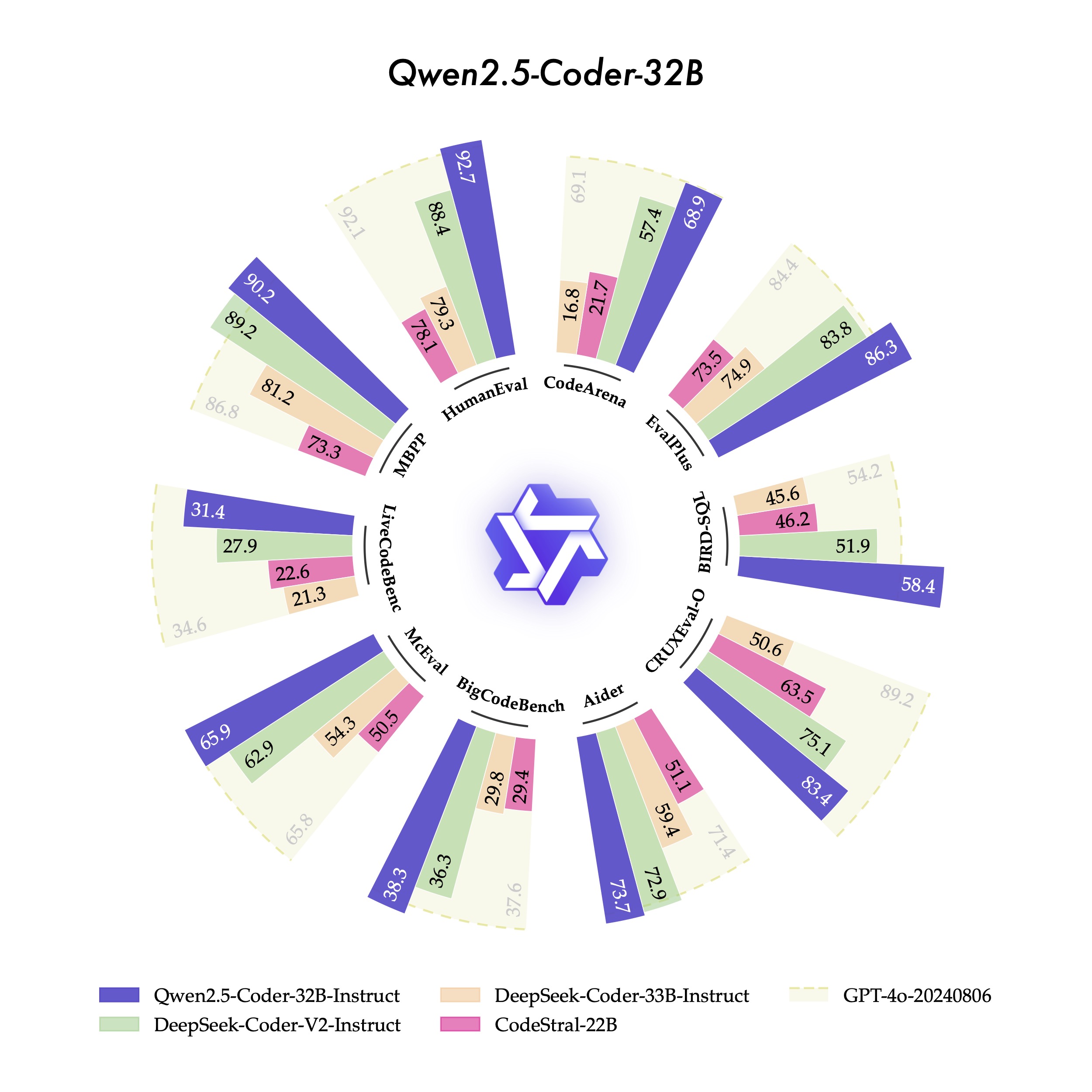
Qwen2.5-Coder 是由阿里云 Qwen 团队开发的大型语言模型系列 Qwen2.5 的代码版本。
- 强大:Qwen2.5-Coder-32B-Instruct 成为当前 SOTA 开源代码模型,具备与 GPT-4o 相匹配的编码能力。
- 多样:提供四种不同规模的模型(0.5B / 3B / 14B / 32B),满足不同开发者的需求。
- 实用:在代码助手和实际应用场景中展示了其潜在应用价值。
- 支持长上下文理解和生成,最大上下文长度达到 128K 个标记。
- 支持 92 种编程语言,并保留基础模型在数学和通用能力上的优势。
iterative/datachain


datachain 是一个现代的 Python 数据框架库,旨在为人工智能提供数据仓库解决方案。 该项目主要解决了如何高效地从云存储中获取、转换和分析非结构化数据的问题。
- 作为真实来源的存储:处理来自 S3、GCP、Azure 和本地文件系统的非结构化数据,无需冗余副本。
- 友好的 Python 数据管道:操作 Python 对象及其字段,内置并行计算和超出内存限制的计算。
- 数据丰富与处理:使用本地 AI 模型生成元数据,通过向量嵌入进行搜索,并将数据集传递给 Pytorch 和 Tensorflow。
- 效率高:支持并行化、超出内存工作负载和数据缓存,对 Python 对象字段进行优化向量操作。
PaddlePaddle/PaddleHelix



PaddleHelix 是生物计算平台,具有大规模表示学习和多任务深度学习功能。
- 提供 HelixFold3 服务器,用于生物分子结构预测
- 开源 HelixFold3 代码和模型参数,实现与 AlphaFold3 相媲美的准确性
- 发布 HelixDock 代码,提高蛋白质配体结构预测准确性
- 接受 Nature Machine Intelligence 论文 “Multi-purpose RNA Language Modeling with Motif-aware Pre-training and Type-guided Fine-tuning”
- 发布技术报告 HelixFold-Multimer,在抗原抗体和肽蛋白结构预测中取得显著成功
- 接受 Nature Machine Intelligence 论文 “A method for multiple-sequence-alignment-free protein structure prediction using a protein language model”
- 发表 BIBM 2022 论文 “HelixMO: Sample-Efficient Molecular Optimization in Scene-Sensitive Latent Space” 并部署药物设计服务
- 公开发布了排名 OGB PCQM4Mv2 榜首的全新分子属性预测网络 HelixGEM -2 的代码
ymcui/Chinese-LLaMA-Alpaca-3



Chinese-LLaMA-Alpaca-3 是基于 Meta Llama 3 开发的中文开源大模型项目。
- 开源了 Llama-3-Chinese 基座模型和指令精调模型,支持多种使用场景。
- 提供预训练和指令精调脚本,便于用户进一步训练或微调。
- 支持在个人电脑上快速进行大模型量化与部署,并提供详细教程。
- 使用大规模中文数据进行增量预训练,提高了基础语义理解能力及指令处理能力。
- 与🤗transformers 等多个生态系统兼容,增强了可用性与灵活性。





