ghostty-org/ghostty



Ghostty 是一个快速、功能丰富且跨平台的终端模拟器,采用平台原生用户界面和 GPU 加速。
- 快速、高效的性能,与其他顶级终端模拟器竞争
- 支持标准兼容性,确保与现有 shell 和软件兼容
- 提供多窗口、标签页和窗格等丰富的窗口功能
- 具备基本自定义选项,如字体和背景颜色设置
- 利用现代技术推动终端模拟器的新特性,为 CLI 工具开发者提供更多可能性
novitalabs/AnimateAnyone



AnimateAnyone 是一个非官方的 Animate Anyone 实现,提供了预训练权重和推理代码。
- 提供非官方的预训练权重和推理代码
- 基于 MooreThreads/Moore-AnimateAnyone 的实现进行调整
- 支持将原始视频转换为姿态视频(关键点序列)
- 兼容 Python 3.10 及以上版本和 CUDA 11.7
- 可通过命令行轻松运行推理脚本
mustafaaljadery/gemma-2B-10M


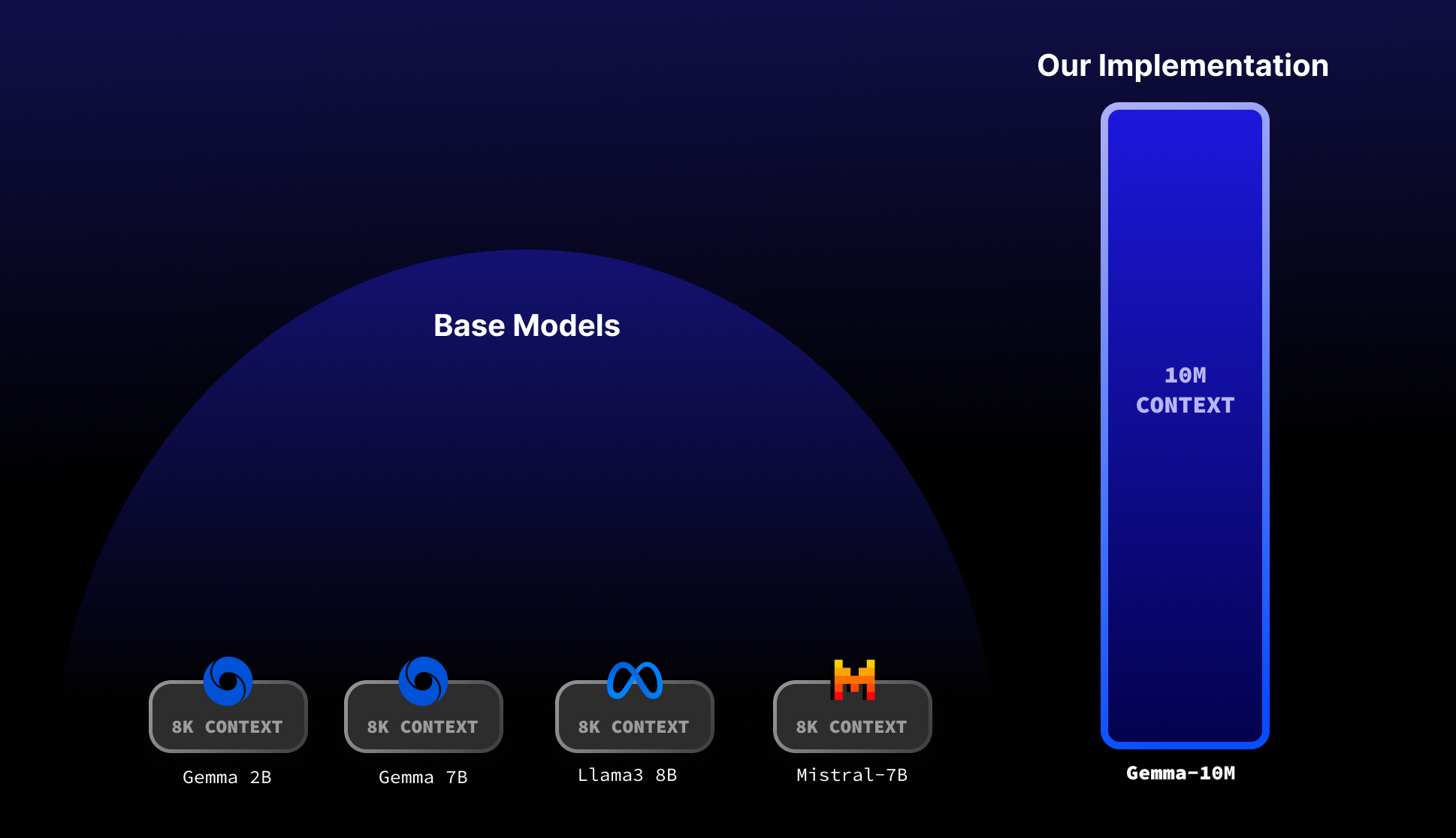
gemma-2B-10M 是一个使用 Infini-attention 的 Gemma 2B 模型,支持长达 10M 的上下文长度。
- 支持高达 10M 的序列长度。
- 在低于 32GB 内存下运行。
- 原生推理优化为 CUDA。
- 使用递归局部注意力实现 O(N) 内存效率。
cisagov/vulnrichment


vulnrichment 是一个用于进行漏洞丰富化的公共存储库。
- 通过 CISA 的 ADP 容器对公共 CVE 记录进行丰富化
- 对新近和现有 CVE 进行评估,并添加关键 SSVC 决策点
- 为高风险 CVE 提供 CWE、CVSS 和 CPE 数据点的补充信息
- 支持使用 GitHub API 和 CVE 服务 API 获取数据
- 不会覆盖原始 CNA 在 CVE 记录中的数据
kyegomez/AlphaFold3


AlphaFold3 是一个基于 PyTorch 的生物分子相互作用结构预测的实现,源自论文“Accurate structure prediction of biomolecular interactions with AlphaFold3”。
- 提供准确的生物分子相互作用结构预测
- 使用 PyTorch 实现,易于集成和扩展
- 支持随机输入生成和模型训练
- 包含 Docker 镜像以简化环境设置
- 采用先进的遗传扩散模块进行坐标处理





